National Cancer Institute, Unsplash
Alexis Verger, Université de Lille
CRISPR-Cas9, c’est la possibilité de couper des séquences d’ADN où on le veut.
Cela peut être très utile par exemple lorsqu’une maladie génétique est causée par une mutation délétère, c’est-à-dire quand une petite partie d’une double hélice d’ADN est mal encodée, et que l’on connaît bien quelle est cette mutation et où elle se trouve. On peut alors, grâce à CRISPR-Cas9, couper le morceau d’ADN impliqué. L’idée est ensuite de remplacer cette séquence mutée par une séquence d’ADN qui corrige la mutation. Ces maladies génétiques causées par une seule mutation sont en fait assez rares, car la plupart d’entre elles sont « multifactorielles » – plusieurs gènes et mutations sont impliqués – mais cela marche plutôt bien. L’utilisation de l’édition des génomes est très encadrée, et la France est signataire de la convention d’Oviedo qui interdit de faire des modifications transmissibles à la descendance.
Pour couper où on veut, il faut d’abord se repérer dans le génome, c’est-à-dire trouver la séquence d’ADN que l’on veut couper. C’est le travail de la première composante de CRISPR-Cas9, que l’on appelle l’ARN-guide (constitué d’une séquence variable complémentaire de la séquence d’ADN à cibler et d’un petit ARN propre à l’enzyme Cas9). Une fois guidée au bon endroit, l’enzyme Cas9 coupe le double brin d’ADN.
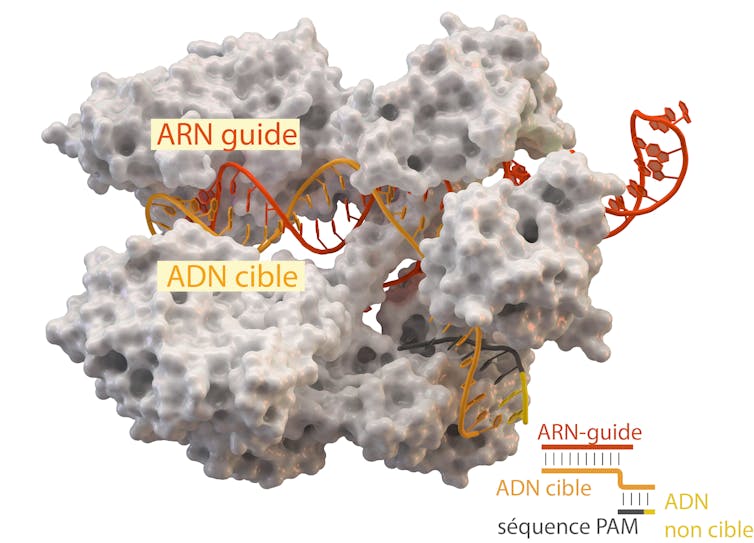
Modifié par Elsa Couderc à partir de Thomas Splettstoesser, Wikipedia, CC BY-SA
On peut faire une analogie avec un traitement de texte : on repère un mot avec une faute d’orthographe au sein d’un énorme ouvrage (l’ARN-guide correspond à la fonction « rechercher »), on coupe le mot erroné (l’enzyme nucléase Cas9 correspond à la fonction « couper »). Ensuite, mais on sort du système CRISPR-Cas9 à proprement parler, on peut ajouter le mot corrigé (la fonction « coller », que nous verrons plus loin).
La force de CRISPR-Cas9 par rapport aux systèmes de découpe du génome que l’on connaissait auparavant, c’est que cette technique est programmable : on peut concevoir et synthétiser la séquence d’ARN-guide correspondant précisément à la séquence d’ADN que l’on souhaite découper. D’autres techniques de découpage du génome existaient avant CRISPR-Cas9, par exemple les « nucléases à doigts de zinc » ou les TALEN, mais elles étaient bien plus complexes et longues à mettre en œuvre.
Le système CRISPR-Cas9 est un système naturel chez les bactéries qui l’utilisent comme mécanisme de défense contre les ADN étrangers. Il a été découvert dans les années 2000 puis « détourné » en 2012 par Jennifer Doudna et Emmanuelle Charpentier pour en faire un outil de biotechnologie et aujourd’hui de thérapie génique.
Comment l’ARN-guide trouve-t-il la séquence qui lui correspond dans tout cet ADN ?
Le complexe formé par l’enzyme Cas9 et son ARN-guide scanne tout au long des chromosomes et s’arrête quand elle trouve la séquence qui correspond à l’ARN-guide. Une des choses fascinantes dans cette technique CRISPR-Cas9 – en dehors de sa facilité de mise en œuvre –, c’est qu’elle est issue d’un système naturel bactérien, mais qu’elle fonctionne bien aussi chez les eucaryotes. En effet, chez ses derniers, la présence d’histones, les principaux constituants protéiques des chromosomes qui n’ont pas d’équivalents chez les bactéries, et d’un noyau ne semblent pas outre mesure empêcher l’enzyme Cas9 d’accéder au génome des eucaryotes et de couper l’ADN.
L’ARN-guide emmène son enzyme Cas9 vers une petite séquence spécifique de 20 nucléotides, alors que le génome humain fait environ 3 milliards de paires de bases au total. L’ARN-guide est complémentaire de la séquence d’ADN que l’on cherche à couper. Par exemple, une séquence ADN-cible ACGGACCTTACCTGAAAAAT est complémentaire d’une séquence TGCCTGGAATGGACTTTTTA (A correspond à T et C à G). La probabilité pour retrouver plusieurs fois la même séquence précise de 20 paires de bases parmi tout le génome est très très faible – même s’il y a encore beaucoup de questionnements à l’heure actuelle sur le risque de couper à un autre endroit que la séquence voulue.
Une fois la cible détectée, l’ARN-guide et l’ADN-cible forment un « duplex » ARN-ADN. L’ARN et l’ADN sont très proches physiquement, et la protéine Cas9 est à portée de ciseaux. Mais elle ne pourra couper le double brin d’ADN-cible que si celui-ci contient également une séquence dite PAM (pour Protospacer Adjacent Motif), constituée de trois nucléotides. Dans le cas de Cas9, le « PAM » est constitué des nucléotides NGG (n’importe quel nucléotide, puis deux G à la suite). Si l’ADN cible ne présente pas cette séquence, l’enzyme Cas9 ne peut pas couper, ce qui restreint bien sûr les applications.
Comment ça coupe ?
Cas9 est une nucléase, c’est-à-dire une enzyme qui clive les liaisons moléculaires reliant les nucléotides de l’ADN. Elle contient deux domaines (RuvC et HNH) qui lui permettent de couper les deux brins d’ADN. Il existe des dizaines d’autres enzymes nucléases en dehors de l’enzyme Cas9.
Après la coupure
Une fois que le système CRISPR-Cas9 a coupé la double hélice d’ADN, la cellule va tout mettre en place pour réparer cette lésion, en raboutant les deux morceaux qui viennent d’être coupés par des mécanismes de réparation cellulaire. La cellule peut soit réparer par la jonction d’extrémités non homologues (NHEJ), ce qui induit des délétions et donc l’inactivation du gène, soit par recombinaison homologue (en présence d’un brin ADN donneur), ce qui permet la réparation.
C’est à l’heure actuelle un sujet important de recherche, car il est toujours difficile d’avoir des réparations efficaces ou qui correspondent à ce que l’on souhaiterait. Par exemple, quand on essaye de corriger une mutation dans des lignées cellulaires au laboratoire, il y a souvent moins d’un pour cent des cellules testées qui ont exactement la bonne coupure et la bonne réparation. Ceci dit, ces problèmes se poseraient quel que soit le mécanisme de découpage utilisé (CRISPR-Cas9, nucléases à doigt de zinc, etc.) puisqu’ils dépendent en fait de la cellule et des mécanismes de réparation cellulaire.
Les « autres » CRISPR – ou comment la technique évolue
Pour les thérapies géniques, il faut d’abord pouvoir accéder à l’ADN incriminé. Il est par exemple plus facile d’accéder au sang et donc à une mutation localisée dans le sang qu’à des organes internes comme le foie. Pour arriver jusqu’à l’ADN, il faut un « véhicule », mais certains de ces transporteurs ne sont pas assez gros pour contenir l’enzyme Cas9, qui est très grande. C’est pour cela que des chercheurs cherchent d’autres enzymes Cas, plus courtes, qui pourront être plus facilement transportées par ces véhicules.
D’autres enzymes Cas sont aussi à l’étude pour leur spécificité de séquence, en particulier pour identifier d’autres séquences PAM, qui permettent la découpe, que la séquence très spécifique NGG associée à l’enzyme Cas9. Ces autres enzymes Cas seront utiles pour cibler les mutations génétiques entourées d’une autre séquence que NGG.
Enfin, des enzymes Cas dites « haute fidélité » permettent de gagner en efficacité, et d’éviter de couper à des endroits non souhaités.![]()
Alexis Verger, Chargé de Recherche au CNRS, ERL 9002, Université de Lille
Cet article est republié à partir de The Conversation sous licence Creative Commons. Lire l’article original.


